La machine learning au service de l’identification des ruminants en contexte archéologique
L'identification des animaux en contexte archéologique s'avère essentielle pour documenter la biodiversité animale passée et les relations entre les humains et les animaux sauvages et domestiques. Une nouvelle étude publiée dans Journal of Computer Applications in Archaeology met en lumière l'intérêt des méthodes de topologie computationnelle pour extraire des informations biologiques fiables et interprétables par les archéozoologues. Cette approche innovante offre l’accès à des données inédites non accessibles par les critères anatomiques traditionnels.
En résumé
- La morphologie squelettique des ruminants domestiques et sauvages s'avère très similaire, rendant leur identification en contexte archéologique relativement difficile.
- A l'inverse, les caractéristiques topologiques se révèlent très discriminantes pour classer les taxons sauvages et identifier les races ovines et caprines domestiques.
- Une étude explore le potentiel de l'analyse topologique des données et de l'apprentissage par noyaux multiples pour identifier spécifiquement les ossements 3D.
Dans de nombreux sites archéologiques d'Asie du Sud-Ouest datant de l'Holocène, les vestiges fauniques comprennent souvent des ossements de divers ongulés sauvages et domestiques, tels que des moutons, des chèvres, des bouquetins, des chevreuils et des gazelles. Ces assemblages fournissent des informations sur les stratégies de chasse et d'élevage, ainsi que des données paléoécologiques sur les sociétés humaines d'autrefois. Cependant, la morphologie très similaire des squelettes rend l'identification difficile à partir des seuls restes osseux.
Pour répondre à cette problématique, une équipe scientifique internationale, composée notamment de chercheurs du laboratoire Cultures et environnements, Préhistoire, Antiquité, Moyen Âge (CEPAM – CNRS / Univ. Côte d’Azur), présente une étude de cas visant à tester le potentiel de l'analyse topologique des données (TDA) et de l'apprentissage par noyaux multiples (MKL). L’objectif de ces méthodes de machine learning est d’évaluer leur efficacité pour l'identification interspécifique de 150 talus [os du pied] 3D, appartenant à des ruminants modernes et anciens provenant de sites archéologiques.
L'application conjointe de ces deux techniques se révèle très efficace pour identifier précisément les espèces sauvages, avec un taux de réussite d'environ 90 %. En revanche, les résultats s'avèrent plus modestes en ce qui concerne les espèces domestiques, en particulier les espèces ovines et caprines, avec un taux d’identification autour de 60 %. Ce faible taux s’explique par la variabilité morphologique de ces races domestiques.
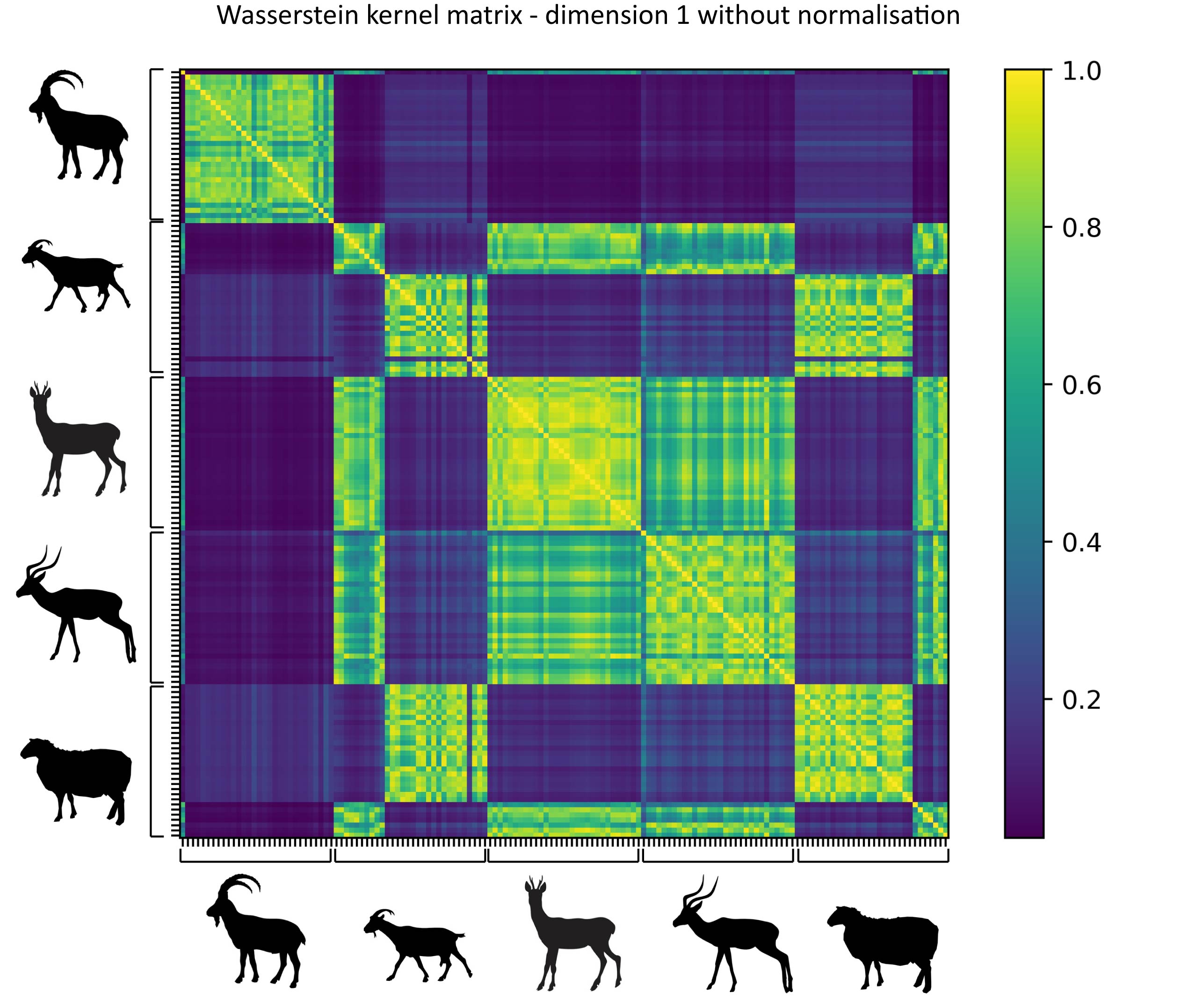
Cette approche innovante offre ainsi un outil prometteur pour étudier la variabilité intraspécifique chez les animaux domestiques et sauvages, ainsi que de nouvelles perspectives pour comprendre les pratiques et techniques de sélection des animaux domestiques du passé, et faciliter le suivi de l'évolution morphologique des populations au fil du temps.
Crédit photo haut de page : © Cyril FRESILLON / TRACES / CNRS Images
Laboratoires CNRS impliqués
- Cultures et environnements, Préhistoire, Antiquité, Moyen Âge (CEPAM – CNRS / Univ. Côte d’Azur)
- Bioarchéologie Interactions Sociétés Environnements (BioArch - CNRS / MNHN / INRAP)
- Environnements et sociétés de l'orient ancien (Archéorient – CNRS / Univ. Lumière Lyon 2)
Référence de la publication
Vuillien, M., Adamo, D., Vila, E., Amane, A., Argant, T., Helmer, D., Mashkour, M., Moussous, A., Notter, O., Rossoni-Notter, E., Théry-Parisot, I., & Corneli, M. (2025). Topological Data Analysis and Multiple Kernel Learning for Species Identification of Modern and Archaeological Small Ruminants. Journal Of Computer Applications In Archaeology, 8(1). Publié le 23 mai 2025.