Un modèle pour prédire la valeur sélective des hybrides
L'hybridation est couramment observée dans la nature et elle est souvent réalisée au laboratoire dans les recherches en génétique et en agronomie pour améliorer les variétés aussi bien animales que végétales. Malgré de nombreux résultats empiriques sur l'hybridation montrant des issues très variées, allant de la vigueur à la non viabilité des hybrides, nous ne possédons pourtant que très peu de prédictions théoriques, et elles sont restreintes à des cas particuliers. Deux chercheurs de l’ISEM en collaboration avec un chercheur de l’Université de Cambridge viennent de publier dans la revue Evolution Letters un modèle mathématique simple et général qui présente une très bonne adéquation avec de nombreux résultats empiriques.
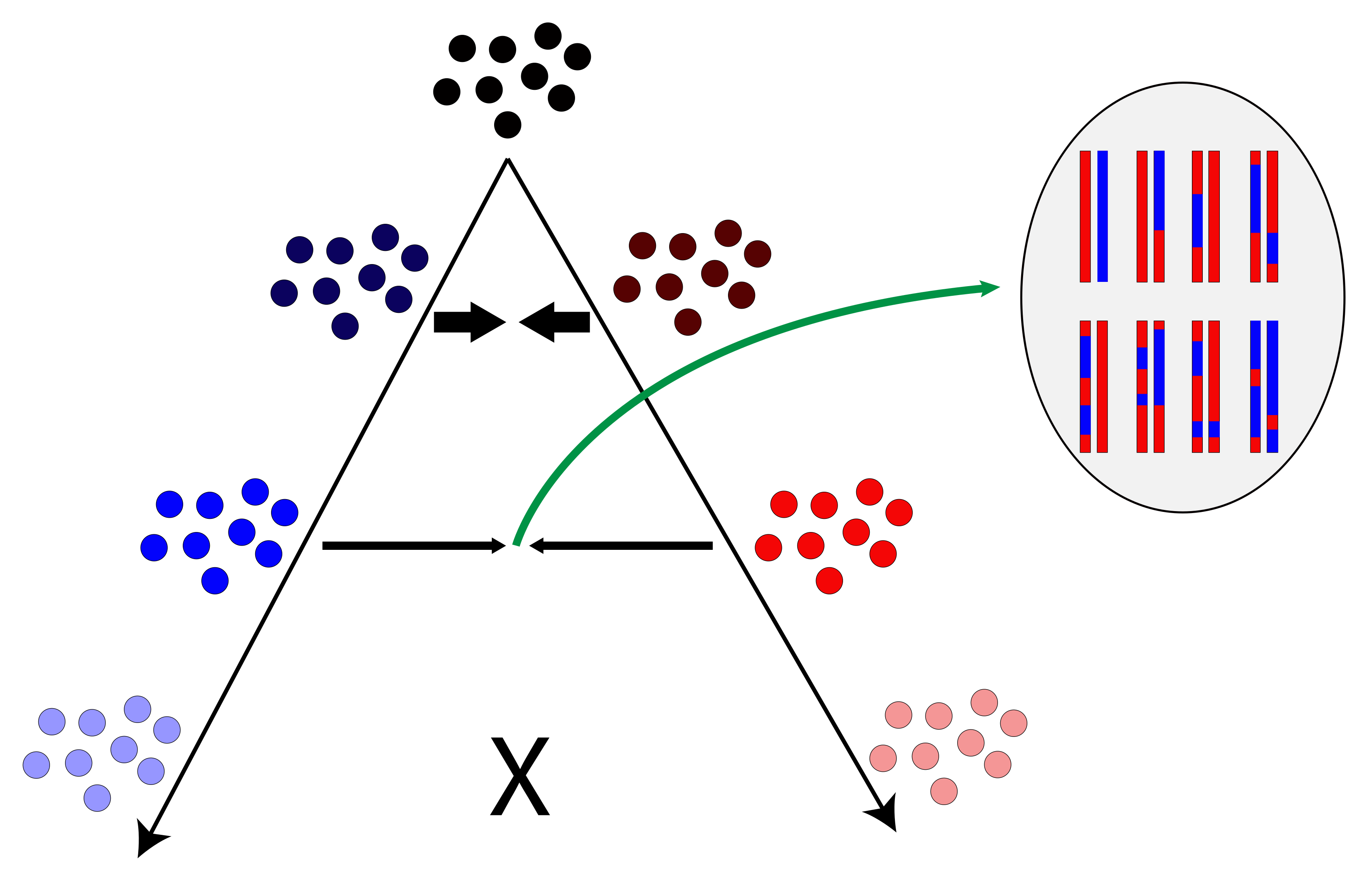
La spéciation correspond à un arrêt progressif de l'hybridation entre lignées (voir figure). En effet, l'hybridation entre populations ou espèces reste possible sur une large gamme de temps et de divergence moléculaire. Il n'est donc pas surprenant d'observer couramment des événements d'hybridation dans la nature ou de pouvoir les réaliser au laboratoire. Comprendre et prédire le résultat de ces hybridations, c'est à dire la valeur sélective des hybrides, est ainsi un champ de recherche majeur en biologie évolutive. Le résultat de l'hybridation a toujours fasciné les chercheurs. Parfois les hybrides montrent de meilleures performances que leurs parents, c'est l'hétérosis ou vigueur hybride. D'autres fois les hybrides présentent une baisse prononcée de fécondité ou de viabilité, c'est la dépression d'hybridation. Le plus souvent les hybrides dévoilent une diversité phénotypique surpassant celle observée dans les populations parentales, c'est ce qu'on appelle la ségrégation transgressive.
Un génotype hybride correspond à une nouvelle combinaison d'allèles qui n'a pas été testée par la sélection naturelle au cours de la divergence. L'issue de l'hybridation dépend donc de la manière dont les allèles interagissent au sein des génomes et pose un problème combinatoire complexe lorsque le nombre de locus impliqués augmente. De ce fait on utilise souvent l’interaction des allèles à un seul locus - l'effet des mutations délétères récessives qui explique plutôt bien la vigueur hybride - ou entre paire de locus - les incompatibilités génétiques dites de Dobzhansky-Muller qui explique plutôt bien la dépression d'hybridation. On peut saluer le succès de ces modèles, cependant leur travers a été d'avoir encouragé l'émergence de la notion de "gène de spéciation", l'idée que la valeur sélective des hybrides est principalement déterminée par un petit nombre de gènes à effet fort. Or rien ne permet de négliger l'impact cumulé des très nombreux gènes à effet plus faible.
Les auteurs de cette nouvelle étude ont pris le problème sous un autre angle. Pour prédire l’impact de la composition génomique des hybrides sur leur valeur sélective, les chercheurs ont utilisé le modèle géométrique de Fisher qui permet de considérer une multitude de sites dans le génome tout en ayant un moyen simple de déterminer la valeur sélective des individus. Plutôt que d'essayer d'identifier (de cartographier) les gènes à effet majeur, ils se sont intéressés à des paramètres globaux de la composition des génomes (proportions provenant de chaque lignée parentale, hétérozygotie) pour comparer les attendus entre et au sein de différentes classes d’hybrides. Le modèle produit des prédictions simples à partir d’un nombre restreint de paramètres facilement accessibles dans de nombreuses études de génomique des populations. Les prédictions sont en accord avec une gamme très large de résultats empiriques, tant chez les animaux que chez les plantes. Le modèle permet de plus d’expliquer des résultats où l'interprétation des processus avait été difficiles par le passé avec les modèles précédents (par ex. des différences entre F2 et backcross ou entre sexes). Enfin, les auteurs démontrent que les modèles d’incompatibilités peuvent donner des résultats similaires à cette approche lorsqu’un jeu de paramètres précis est sélectionné, permettant de rapprocher les deux cadres théoriques.
Ce nouveau modèle est une approche multigénique, complémentaire des approches se basant sur les “gènes de spéciation” à fort effet, qui pourra permettre d'interpréter sous un nouvel angle bon nombre de données de génomique de la spéciation.
Simon A., Bierne N., Welch J. J. Coadapted genomes and selection on hybrids: Fisher's geometric model explains a variety of empirical patterns. Evolution Letters. doi: 10.1002/evl3.66