Modélisation de la propagation des variants en France : de l’émergence au remplacement
Les variants du SARS-CoV-2 ont radicalement changé la donne de l’épidémie de COVID-19, conduisant par exemple à un 3e confinement national. Une collaboration née à l’automne 2020 entre l’équipe de modélisation « Évolution Théorique et Expérimentale » du laboratoire Maladies infectieuses et vecteurs : écologie, génétique, évolution et contrôle (MIVEGEC – CNRS / IRD / Université de Montpellier), le CHU de Montpellier et le laboratoire CERBA a rapidement permis de quantifier l’avantage de transmission des variants et de suivre leur propagation. Une partie de ces travaux a été publiée dans les revues Emerging Infectious Diseases et Eurosurveillance.
La fin de l’année 2020 a vu l’émergence de 3 variants préoccupants, désormais appelés α, β et γ, détectés respectivement au Royaume-Uni, en Afrique-du-Sud et au Brésil. De par leur propriétés infectiologiques différentes, ils ont été à l’origine de rebonds épidémiques majeurs dans ces trois pays.
Dès la mi-janvier 2021, les analyses des équipes britanniques ont montré que le variant α (issu de la lignée B.1.1.7) était plus contagieux. Des tests dits de criblage ont alors été mis en place sur le territoire français. L’idée était de re-tester tous les positifs afin de déterminer si l’infection avait été causée par l’un des trois variants préoccupants.
Grâce à une collaboration et une étude clinique mise en place dès la fin 2020 entre l’équipe de modélisation ETE du laboratoire Maladies infectieuses et vecteurs : écologie, génétique, évolution et contrôle (MIVEGEC – CNRS / IRD / Université de Montpellier), le CHU de Montpellier et le laboratoire CERBA, les chercheurs ont pu analyser les résultats des tests de criblage en France et partager dès le 23 février 2021 la première estimation de l’avantage de transmission des variants en France par rapport aux souches ancestrales. Celui-ci s’élevait à 50 % dans un scénario pessimiste et à 38 % dans un scénario conservateur avec une forte hétérogénéité régionale. Pour cela, ils ont appliqué des modèles de génétique des populations adaptés à la quantification de la propagation d’un allèle mutant dans une population résidente. Ces travaux ont été publié le 5 mai dans la revue du Centers for Diseases Control Emerging Infectious Diseases.
La propagation du variant α a été telle qu’en avril de nouveaux tests de criblage ont été mis en place afin de mieux détecter les deux autres variants β et γ, dont les infections semblent échapper à l’immunité naturelle. En combinant les tests de criblage avec des données de séquençage des génomes viraux, les analyses du même consortium ont montré qu’au mois d’avril ces variants β et γ ont pris le dessus sur le variant α dans plusieurs régions dont l’Île-de-France et les Hauts-de-France. Ces derniers résultats ont des implications pour la gestion de l’épidémie de COVID-19 en France car l’évasion immunitaire pourrait nécessiter des mesures de contrôle renforcées. Ces résultats sont à paraître dans la revue Eurosurveillance, la revue du centre européen de prévention et de contrôle des maladies.
Ces travaux illustrent le potentiel des modèles issus de la dynamique des populations en santé publique, en particulier pour décrire, comprendre et anticiper la propagation et l’évolution virale.
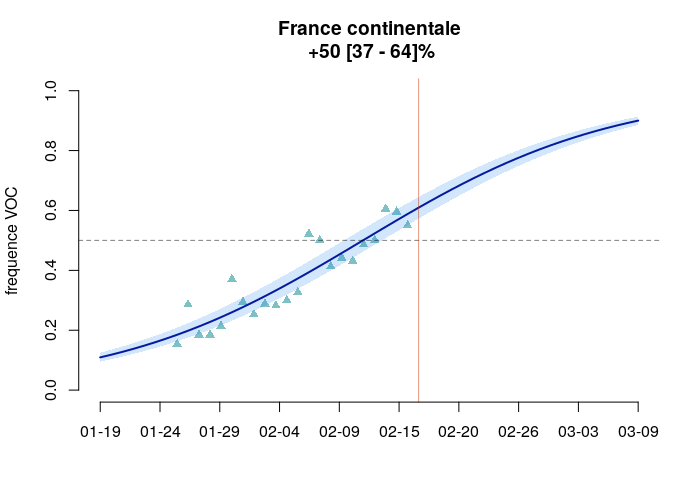
Figure basée sur les données de Haim-Boukobza et al et représentant la proportion de variants préoccupants (VOC) en France inférée par le modèle (la courbe bleue) à partir des observations (les triangles).
©Adobestock / CERBA Health Care
Références
Haim-Boukobza S, Roquebert B, Trombert-Paolantoni S, Lecorche E, Verdurme L, Foulongne V, Selinger C, Michalakis Y, Sofonea MT, Alizon S. 2021. Detecting rapid spread of SARS-CoV-2 Variants, France, January 26–February 16, 2021. Emerging Infectious Diseases 27:1496–1499.
Roquebert B, Trombert-Paolantoni S, Haim-Boukobza S, Lecorche E, Verdurme L, Foulongne V, Sofonea MT, Alizon S. 2021. The SARS-CoV-2 B.1.351/V2 variant is outgrowing the B.1.1.7/V1 variant in French regions in April 2021. Eurosurveillance, in press
Les objectifs de développemenht durable

- ODD 3 - Santé et bien-être